mychisqunif = function(n=3,N=1000) {
sample = runif(N,-1,1)
if (n >1) {
for (i in 1:(n-1)) {
sample = sample + runif(N,-1,1)
}
}
sigmacentral=2*sqrt(n/12)
delta=sigmacentral/20
h=hist(sample,breaks=seq(-1*n,1*n,delta))
curve(dnorm(x,sd=sigmacentral)*N*delta,add=T,col="red")
x=h$mids
dati=h$counts
attesi = dnorm(x,sd=sigmacentral)*N*delta
chi2 = sum((dati-attesi)^2/attesi)
ngl = length(x) - 1
cat("chi2= ",chi2," dof=",ngl," p-value is ",1-pchisq(chi2,df=ngl),"\n")
}
|
Con i valori ![]() ,
, ![]() , il test dà tipicamente valori del
p-value alti che non ci permettono di escludere l'ipotesi che il
campione segua la distribuzione normale. Per poter ``accorgersi'' dai
dati che la reale distribuzione non è una gaussiana, è necessario
disporre di statistiche molto superiori:
, il test dà tipicamente valori del
p-value alti che non ci permettono di escludere l'ipotesi che il
campione segua la distribuzione normale. Per poter ``accorgersi'' dai
dati che la reale distribuzione non è una gaussiana, è necessario
disporre di statistiche molto superiori:
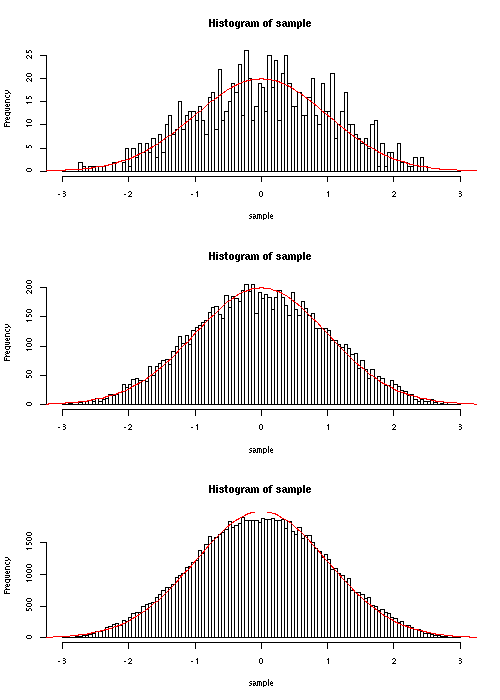
> par(mfrow=c(3,1)) > mychisqunif(3,1000) chi2= 138.0771 dof= 119 p-value is 0.1114694 > mychisqunif(3,10000) chi2= 157.4705 dof= 119 p-value is 0.01048201 > mychisqunif(3,100000) chi2= 703.2427 dof= 119 p-value is 0 |
chisqcosmic = function() {
df = read.table(file="CosmoData.txt",skip=7)
c = df$V3
cm=mean(c)
n=length(c)
# costruiamo l'istogramma con bins di larghezza 1 centrati nei valori interi
xmin=min(c)-1.5
xmax=max(c)+1.5
x=xmin:xmax
h=hist(c,breaks=x)
xp=h$mids
dati=h$counts
attesi = dpois(xp,lambda=cm)*n
points(xp,attesi,type="p",col="red",pch=19)
chi2 = sum((dati-attesi)^2/attesi)
nbin=length(x)
ngl = nbin - 2
cat("chi2= ",chi2," dof=",ngl," p-value is ",1-pchisq(chi2,df=ngl),"\n")
}
|
Si noti che in questo caso il numero di gradi di libertà è N-2, poichè abbiamo usato i dati, oltre che per normalizzare la predizione, anche per stimarne il valore atteso.
Dal risultato p-value=65% concludiamo che i dati sono del tutto compatibili con l'ipotesi poissoniana.

Contando la frequenza delle osservazioni in intervalli di 2 m, si
osserva un possibile segnale intorno al valore ![]() m :
m :
misure = scan("/afs/math.unifi.it/service/Rdsets/ricercasegnale.rdata")
h=hist(misure,breaks=seq(0,100,2))
|
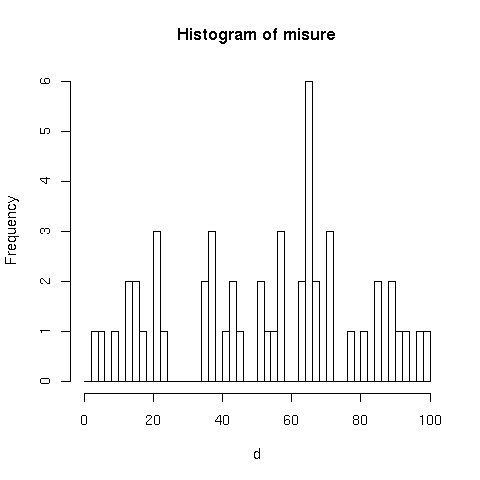
Dai conteggi in istogramma possiamo calcolare il ![]() :
:
> dati=h$counts
> ngl = length(dati)
> fondo.atteso=1.04 # per bin
> attesi=rep(fondo.atteso,ngl)
> chi2 = sum((dati-attesi)^2/attesi)
> cat("chi2= ",chi2," dof=",ngl," p-value is ",1-pchisq(chi2,df=ngl),"\n")
chi2= 68.26923 dof= 50 p-value is 0.0438842
|
Essendo il p-value ottenuto inferiore ad ![]() , potremmo
concludere che i dati non sono compatibili con un fondo
uniforme. Tuttavia, dato il basso valore dei conteggi, non possiamo
aspettarci che la distribuzione di Pearson sia una buona
approssimazione per la distribuzione del nostro
, potremmo
concludere che i dati non sono compatibili con un fondo
uniforme. Tuttavia, dato il basso valore dei conteggi, non possiamo
aspettarci che la distribuzione di Pearson sia una buona
approssimazione per la distribuzione del nostro ![]() . Nel caso
di bassi (
. Nel caso
di bassi (
![]() ) conteggi, la distribuzione del
) conteggi, la distribuzione del ![]() dipende dal caso in esame, e per calcolare il valore
corretto del p-value è necessario determinare la distribuzione
corretta del
dipende dal caso in esame, e per calcolare il valore
corretto del p-value è necessario determinare la distribuzione
corretta del ![]() simulando un gran numero di
analoghi esperimenti nell'ipotesi nulla.
simulando un gran numero di
analoghi esperimenti nell'ipotesi nulla.
lowsignal = function(Nsim=5000) {
misure = scan("/afs/math.unifi.it/service/Rdsets/ricercasegnale.rdata")
h=hist(misure,breaks=seq(0,100,2))
dati=h$counts
ngl = length(dati)
fondo.atteso=1.04 # per bin
attesi=rep(fondo.atteso,ngl)
# simuliamo Nsim esperimenti con solo fondo e mettiamo nel vettore
# simchi2 i valori del chi2 ottenuto in ciascun esperimento
simchi2=c()
for (i in 1:Nsim) {
simset=rpois(ngl,lambda=fondo.atteso)
simchi2[i]=sum((simset-attesi)^2/attesi)
}
# calcoliamo la frazione di esperimenti simulati con un valore di chi2
# superiore a quello osservato
np=length(simchi2[simchi2>=chi2])
pv=np/Nsim
dpv=sqrt(pv*(1-pv)/Nsim)
cat("correct p-value is ",pv," +/- ",dpv,"\n")
# confrontiamo la distribuzione di chi2 simulata
# con quella di Pearson
ch=hist(simchi2,breaks=50)
delta=ch$breaks[2]-ch$breaks[1]
curve(dchisq(x,df=ngl)*Nsim*delta,add=T,col="red")
}
> lowsignal()
correct p-value is 0.0852 +/- 0.003948188
|
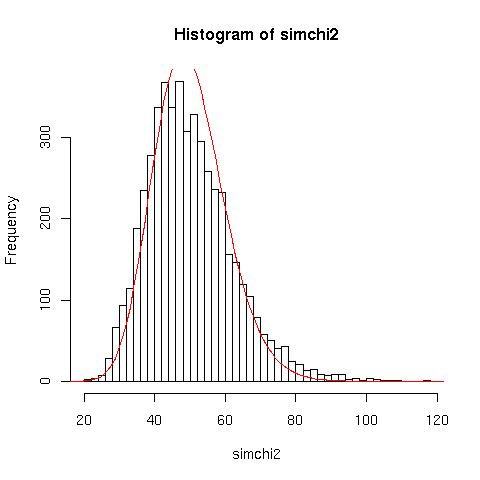
Notiamo che la reale distribuzione del ![]() ha code più importanti
rispetto a quella attesa nel limite asintotico di grandi conteggi, per
cui il valore corretto del p-value è sensibilmente
maggiore di quanto stimato in precedenza.
ha code più importanti
rispetto a quella attesa nel limite asintotico di grandi conteggi, per
cui il valore corretto del p-value è sensibilmente
maggiore di quanto stimato in precedenza.
Concludiamo dunque che entro il limite di significatività dato, i dati sono compatibili con un fondo uniforme.