

da un controllo visuale delle 4 variabili risulta evidente che la specie ``setosa'' può essere isolata dalle altre con un semplice taglio sulla variabile Petal.Length
library(MASS) ldahist(iris$Petal.Length, iris$Species) |
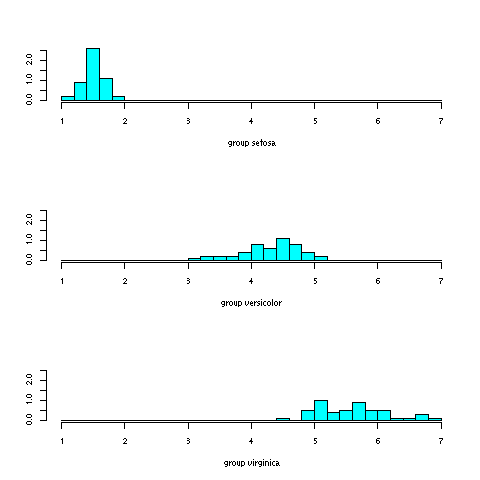
Costruiamo dunque un discriminante lineare per separare l'ipotesi ``versicolor'' dall'ipotesi alternativa ``virginica'':
myiris=iris[iris$Petal.Length>2.5,] set.seed(99) samp=sample(1:100,50) myiris.train=myiris[samp,] myiris.test =myiris[-samp,] # calcolo delle medie nelle due ipotesi mu0 = mean(myiris.train[myiris.train$Species=="versicolor",1:4]) mu1 = mean(myiris.train[myiris.train$Species=="virginica",1:4]) #calcolo delle matirice var/cov nelle due ipotesi v0 = var(myiris.train[myiris.train$Species=="versicolor",1:4]) v1 = var(myiris.train[myiris.train$Species=="virginica",1:4]) #calcolo dei coefficienti lineari a = solve(v0+v1) %*% (mu0-mu1) print(a) |
(si noti l'uso della funzione sample per estrarre casualmente 50 righe dal dataframe, e della funzione set.seed per poter riprodurre la stessa sequenza pseudorandom nel caso di ripetuta esecuzione del codice)
Possiamo a questo punto calcolare la funzione discriminante e osservare la separazione ottenuta sul campione di test:
discr = as.matrix(myiris.test[1:4]) %*% a myiris.test = data.frame(myiris.test,fisher=discr) ldahist(myiris.test$fisher,myiris.test$Species) |
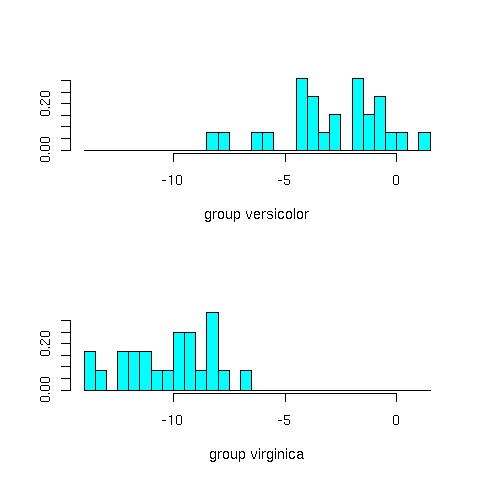
Il discriminante può essere ottenuto in modo equivalente con la
funzione di R lda:
iris.lda2=lda(Species ~ . , myiris.train,prior=c(0,1,1)/2) print(iris.lda2$scaling) |
La funzione lda permette anche l'analisi nel caso di più ipotesi alternative, possiamo dunque usarla direttamente per separare le tre specie:
set.seed(99) samp=sample(1:150,75) myiris3.tr=iris[samp,] myiris3.test=iris[-samp,] iris.lda3=lda(Species ~ . , myiris3.tr, prior=c(1,1,1)/3) print(iris.lda3$scaling) |
Si ottengono in questo caso due statistiche discriminanti; dalla
differenza degli autovalori (
![]() ) possiamo aspettarci che
solo il primo sia significativo, come possiamo verificare
graficamente:
) possiamo aspettarci che
solo il primo sia significativo, come possiamo verificare
graficamente:
a1= iris.lda3$scaling[,1]
a2= iris.lda3$scaling[,2]
myiris3.test = data.frame(myiris3.test, fisher1= as.matrix(myiris3.test[1:4]) %*% a1,
fisher2= as.matrix(myiris3.test[1:4]) %*% a2 )
ldahist(myiris3.test$fisher1,myiris3.test$Species)
ldahist(myiris3.test$fisher2,myiris3.test$Species)
|