
I dati possono essere descritti dal modello lineare
par(mfrow=c(2,1))
cg=read.table("/afs/math.unifi.it/service/Rdsets/cadutalibera.rdata")
attach(cg)
plot(altezza ~ tempo)
n=length(altezza)
# creiamo la matrice del modello
A= matrix( nrow=n,ncol=2,data=c(rep(1,n),-tempo^2/2))
K = solve(t(A) %*% A)
# ricaviamo il vettore theta con la stima dei parametri h0 e g
theta = K %*% t(A) %*% altezza
# disegniamo la funzione ottenuta dal fit
curve(theta[1]-theta[2]*x^2/2,add=T,col="blue")
# stima dell'errore su h
residui = altezza - A %*% theta
plot(residui)
var.h = sum(residui^2)/(n-2)
cat("errore stimato su h:",sqrt(var.h),"\n")
# stima dell'errore sui parametri
V.theta = var.h * K
sigma.theta = sqrt(diag(V.theta))
cat("h0=",theta[1]," +/- ",sigma.theta[1],"\n")
cat("g=",theta[2]," +/- ",sigma.theta[2],"\n")
# h0= 2.097576 +/- 0.01057463
# g= 10.24337 +/- 0.3232533
|
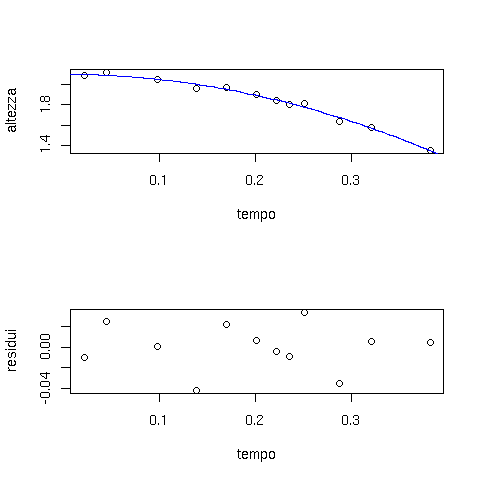
Il grafico che mostra i residui in funzione di ![]() ci permette di
verificare l'assenza di una eventuale dipendenza residua non inclusa nel
modello.
ci permette di
verificare l'assenza di una eventuale dipendenza residua non inclusa nel
modello.
la funzione lm() di R ci permette di svolgere il calcolo con un'unica chiamata:
> summary(lm(altezza ~ I(-tempo^2/2),data=cg))
Call:
lm(formula = altezza ~ I(-tempo^2/2), data = cg)
Residuals:
Min 1Q Median 3Q Max
-0.041615 -0.009098 0.003080 0.010776 0.033711
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 2.09758 0.01057 198.36 < 2e-16 ***
I(-tempo^2/2) 10.24337 0.32325 31.69 2.30e-11 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.02358 on 10 degrees of freedom
Multiple R-Squared: 0.9901, Adjusted R-squared: 0.9892
F-statistic: 1004 on 1 and 10 DF, p-value: 2.303e-11
|
lm(altezza ~ 0 + I(tempo^2)))

Conscendo
![]() m, possiamo ora calcolare il
m, possiamo ora calcolare il ![]() del
fit e il corrispondente p-value:
del
fit e il corrispondente p-value:
sigma.y = 0.03
chi2 = sum(residui^2)/sigma.y^2
cat("chi2=",chi2," con ",n-2," gradi di liberta'\n" )
cat("p-value = ",1-pchisq(chi2,df=n-2),"\n")
|
Il valore ottenuto p-value=0.80 ci conferma la validità del modello.
Gli errori su ![]() possono essere specificati nella funzione
lm() di R tramite l'argomento
possono essere specificati nella funzione
lm() di R tramite l'argomento ![]() che rappresenta il
vettore dei pesi
che rappresenta il
vettore dei pesi
![]() :
:
fit = lm(altezza ~ I(tempo^2/2), weights=rep(1/sigma.y^2,n)) chi2 = deviance(fit) |

La stima di ![]() è ovviamente
è ovviamente
La stima della sua varianza è ottenuta dalla formula di propagazione degli errori, che vale esattamente nel caso lineare:
La variabile
mypred = function(tprime=0.6,alpha=0.95) {
A.tprime= matrix( nrow=1,ncol=2,data=c( 1,-tprime^2/2))
h.pred= A.tprime %*% theta
sigma.pred = sqrt(A.tprime %*% V.theta %*% t(A.tprime))
Nsigma = qt((1+alpha)/2,df=n-2)
cat("h(",tprime,")=",h.pred, " +/- ",Nsigma*sigma.pred," C.L.=",alpha,"\n")
}
> mypred(0.6,0.95)
h( 0.6 )= 0.2537696 +/- 0.1126419 C.L.= 0.95
|
Al solito, R prevede una funzione (![]() ) per fare questo
calcolo in una sola chiamata:
) per fare questo
calcolo in una sola chiamata:
> myfit = lm(altezza ~ I(-tempo^2/2),data=cg)
> predict(myfit,newdata=(tempo=0.6),interval="confidence",level=0.95)
fit lwr upr
[1,] 0.2537696 0.1411276 0.3664115
|

In questo caso ci aspettiamo che i conteggi ![]() misurati in ogni
intervallo
misurati in ogni
intervallo ![]() seguano una distribuzione di Poisson con valore atteso
seguano una distribuzione di Poisson con valore atteso ![]() .
Possiamo dunque dare una prima stima della varianza
.
Possiamo dunque dare una prima stima della varianza
![]() e ottenere un primo fit minimizzando
e ottenere un primo fit minimizzando
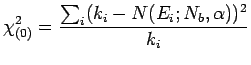
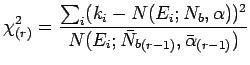
histofit = function() {
sam=scan("/afs/math.unifi.it/service/Rdsets/gaussconfondo.rdata")
iter=0
while (iter<5 ) {
h=hist(sam,breaks=seq(0,10,0.2))
mu=5.2
sigma=0.3
x=h$mids
k=h$counts
n=length(k)
if (iter == 0) {
cat("######### Primo fit #########\n")
dk2=k
}
else {
dk2=A %*% theta
cat("######### Iterazione ",iter," #########\n")
}
# costruiamo la matrice del modello e la matrice var/cov delle k
A = matrix(ncol=2,nrow=N,data= c(rep(1,N) , exp(-(x-mu)^2/(2*sigma^2))))
V.k = matrix(ncol=N,nrow=N,data=0)
diag(V.k) = dk2
V.theta = solve(t(A) %*% solve(V.k) %*% A )
theta = V.theta %*% t(A) %*% solve(V.k) %*% k
cat("theta=",theta,"\n")
cat(" +/- ",sqrt(diag(V.theta)),"\n")
curve( theta[1] + theta[2] *exp(-(x-mu)^2/(2*sigma^2)),0,10,add=T,col="red" )
residui = k - A %*% theta
chisq = sum(residui^2/dk2 )
cat ("chi2=",chisq," con ",N-2," g.d.l. p-value=",1-pchisq(chisq,df=N-2),"\n")
iter=iter+1
}
> histofit()
######### Primo fit #########
theta= 16.91985 156.4992
+/- 0.6252381 7.135217
chi2= 77.57949 con 48 g.d.l. p-value= 0.004377989
######### Iterazione 1 #########
theta= 18.59739 154.8243
+/- 0.6275628 7.171216
chi2= 63.74022 con 48 g.d.l. p-value= 0.06366115
######### Iterazione 2 #########
theta= 18.60706 154.6957
+/- 0.6573149 7.197776
chi2= 58.38637 con 48 g.d.l. p-value= 0.1448092
######### Iterazione 3 #########
theta= 18.60719 154.6940
+/- 0.6574779 7.195656
######### Iterazione 4 #########
theta= 18.60719 154.694
+/- 0.65748 7.195629
chi2= 58.36089 con 48 g.d.l. p-value= 0.1453295
|

Il grafico
attach(MASS) plot(y ~ Ineq, data=UScrime) |
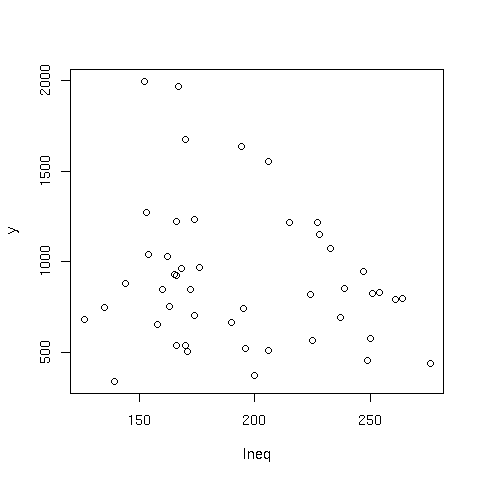
non mostra una evidente correlazione fra le due variabili, anche se
suggerisce una possibile anticorrelazione. Esploriamo dunque il
modello lineare
![]()
> summary(lm(y ~ Ineq, data=UScrime))
Call:
lm(formula = y ~ Ineq, data = UScrime)
Residuals:
Min 1Q Median 3Q Max
-658.54 -271.38 -30.02 183.75 1017.06
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 1241.773 281.478 4.412 6.33e-05 ***
Ineq -1.736 1.422 -1.221 0.229
|
Otteniamo effettivamente un valore negativo per ![]() , tuttavia
il test di Student dell'ipotesi
, tuttavia
il test di Student dell'ipotesi ![]() dà p-value = 23 %,
e non possiamo dunque affermare di aver messo in evidenza una
dipendenza fra le due variabili.
dà p-value = 23 %,
e non possiamo dunque affermare di aver messo in evidenza una
dipendenza fra le due variabili.
Se ora consideriamo la variabile ![]() , notiamo che questa è
evidentemente correlata con le altre due variabili:
, notiamo che questa è
evidentemente correlata con le altre due variabili:
# valutiamo i coefficienti di correlazione lineare
> cor(UScrime[,c("y","Ineq","GDP")])
y Ineq GDP
y 1.0000000 -0.1790237 0.4413199
Ineq -0.1790237 1.0000000 -0.8839973
GDP 0.4413199 -0.8839973 1.0000000
# e il loro errore:
> sqrt( (1-cor(UScrime[,c("y","Ineq","GDP")])^2) / length(UScrime$y))
y Ineq GDP
y 0.0000000 0.14350851 0.13089192
Ineq 0.1435085 0.00000000 0.06819072
GDP 0.1308919 0.06819072 0.00000000
|
Verifichiamo allora una possibile dipendenza di ![]() da
da ![]() dopo
aver tenuto conto della dipendenza da
dopo
aver tenuto conto della dipendenza da ![]() con una semplice ipotesi
lineare:
con una semplice ipotesi
lineare:
![]()
> summary(fit <- lm(y ~ Ineq + GDP, data=UScrime))
Call:
lm(formula = y ~ Ineq + GDP, data = UScrime)
Residuals:
Min 1Q Median 3Q Max
-610.09 -193.09 -16.01 108.05 814.69
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -3639.047 968.692 -3.757 0.000503 ***
Ineq 9.364 2.424 3.863 0.000364 ***
GDP 5.192 1.002 5.179 5.32e-06 ***
|
Vista la forte correlazione fra ![]() e
e ![]() , ci aspettiamo che i
due parametri
, ci aspettiamo che i
due parametri ![]() e
e ![]() siano pure correlati:
siano pure correlati:
> cov2cor(vcov(fit))
(Intercept) Ineq GDP
(Intercept) 1.0000000 -0.9660638 -0.9728075
Ineq -0.9660638 1.0000000 0.8839973
GDP -0.9728075 0.8839973 1.0000000
|
Essendo comunque i due parametri significativamente diversi da 0,
possiamo concludere di aver messo in evidenza una correlazione
positiva fra ![]() e
e ![]() per stati con lo stesso prodotto interno lordo.
Possiamo visualizzare il risultato disegnando l'intervallo di
confidenza per
per stati con lo stesso prodotto interno lordo.
Possiamo visualizzare il risultato disegnando l'intervallo di
confidenza per ![]() e
e ![]() , che nell'ipotesi di errori
gaussiani è definito dai punti all'interno dell'ellisse di covarianza
per cui
, che nell'ipotesi di errori
gaussiani è definito dai punti all'interno dell'ellisse di covarianza
per cui
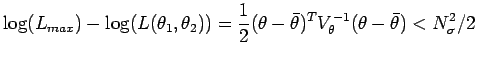
il seguente codice disegna le ellissi corrispondenti a
![]() , che nel caso bidimensionale corrisponde agli intervalli
di confidenza 39.3 %, 86.5 %, 98.9 %
, che nel caso bidimensionale corrisponde agli intervalli
di confidenza 39.3 %, 86.5 %, 98.9 %
mylik = function(t1,t2,theta1,theta2,s1,s2,rho) {
-1/(2*(1-rho)) * ( (t1-theta1)^2/s1^2 +
(t2-theta2)^2/s2^2 -
2*rho* (t1-theta1)/s1 *(t2-theta2)/s2)
}
nsigma=c(1,2,3)
theta1=coef(fit)["Ineq"]
theta2=coef(fit)["GDP"]
s1=sqrt(vcov(fit)["Ineq","Ineq"])
s2= sqrt(vcov(fit)["GDP","GDP"])
rho = cov2cor(vcov(fit))["Ineq","GDP"]
x=seq(-2,15,.1)
y=seq(-2,15,.1)
contour(x,y, outer(x,y,mylik,
theta1=theta1, theta2=theta2,s1=s1,s2=s2,rho=rho),
levels = -nsigma^2/2 , xlab=expression(theta[1]),ylab=expression(theta[2]))
|
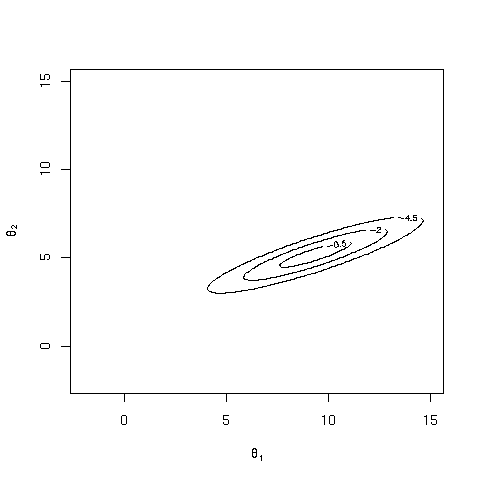
I residui parziali
![]()
![]()
permettono di visualizzare la dipendenza residua da ogni singola
variabile dopo aver tenuto conto delle altre variabili del
fit. Possono essere visualizzati tramite la funzione
![]() di R
di R
termplot(fit,partial=T,se=T) |
Sottolineiamo infine il fatto che i risultati di queste analisi di regressione possono mettere in evidenza le mutue dipendenze fra le variabili, ma non potranno mai dimostrare un rapporto di causa-effetto! Il modello deterministico delle dipendenze è lasciato all'interpretazione dell'analista...

Testiamo l'ipotesi nulla tramite un'analisi della covarianza del
modello M1. Estendiamo cioè l'idea dell'analisi della varianza al caso
in cui la dispersione della variabile studiata può dipendere da una o
più variabili continue tramite ![]() parametri:
parametri:
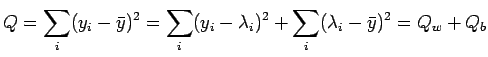
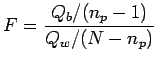
library(MASS)
fit1=lm(loss~hard + tens,data=Rubber)
np=3
N=length(Rubber$loss)
Qb=sum((fit1$fitted.values-mean(Rubber$loss))^2)
Qw=deviance(fit1)
F = Qb/(np-1) / ( Qw/(N-np) )
cat("F= ",F," p-value=",1-pf(F,df1=np-1,df2=N-np),"\n")
|
Il valore del p-value=
![]() dimostra la dipendenza
fra le variabili, escludendo l'ipotesi nulla.
dimostra la dipendenza
fra le variabili, escludendo l'ipotesi nulla.
Si noti che il risultato di questa analisi è riportato anche dalla
chiamata
![]() .
.
Si noti anche che nel caso di un singolo parametro di dipendenza
(retta dei minimi quadrati),
> summary(lm(loss~tens,data=Rubber))
Call:
lm(formula = loss ~ tens, data = Rubber)
Residuals:
Min 1Q Median 3Q Max
-155.640 -59.919 2.795 61.221 183.285
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 305.2248 79.9962 3.815 0.000688 ***
tens -0.7192 0.4347 -1.654 0.109232
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 85.56 on 28 degrees of freedom
Multiple R-Squared: 0.08904, Adjusted R-squared: 0.0565
F-statistic: 2.737 on 1 and 28 DF, p-value: 0.1092
|
Per il confronto fra i modelli M1 e M5, possiamo usare ancora un'analisi di varianza scomponendo i residui del primo fit:
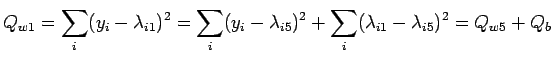
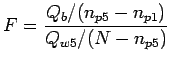
library(MASS)
N=length(Rubber$loss)
fit1=lm(loss~hard + tens,data=Rubber)
np1=3
fit5=lm(loss~hard + tens + I(tens^2) + I(tens^3)+ I(tens^4)+ I(tens^5),data=Rubber)
np5=7
Qb=sum((resid(fit1)-resid(fit5))^2)
Qw5=deviance(fit5)
F = Qb/(np5-np1) / ( Qw5/(N-np5) )
cat("F= ",F," p-value=",1-pf(F,df1=np5-np1,df2=N-np5),"\n")
|
Lo stesso risultato è riprodotto da
> anova(fit1,fit5) Analysis of Variance Table Model 1: loss ~ hard + tens Model 2: loss ~ hard + tens + I(tens^2) + I(tens^3) + I(tens^4) + I(tens^5) Res.Df RSS Df Sum of Sq F Pr(>F) 1 27 35950 2 23 12884 4 23065 10.294 6.286e-05 *** --- Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 |
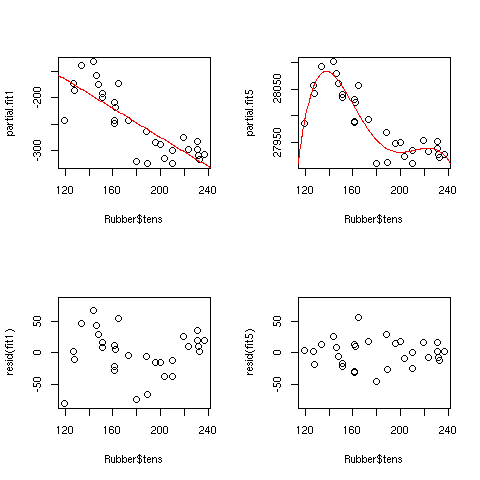
par(mfrow=c(2,2)) library(MASS) N=length(Rubber$loss) fit1=lm(loss~hard + tens,data=Rubber) fit5=lm(loss~hard + tens + I(tens^2) + I(tens^3)+ I(tens^4)+ I(tens^5),data=Rubber) partial.fit1=Rubber$loss - ( coef(fit1)[1] + coef(fit1)[2]*Rubber$hard) partial.fit5=Rubber$loss - ( coef(fit5)[1] + coef(fit5)[2]*Rubber$hard) plot( partial.fit1 ~ Rubber$tens) curve(coef(fit1)[3]*x,add=T,col="red" ) plot( partial.fit5 ~ Rubber$tens) curve(coef(fit5)[3]*x+coef(fit5)[4]*x^2+coef(fit5)[5]*x^3+coef(fit5)[6]*x^4+coef(fit5)[7]*x^5,add=T,col="red" ) plot(resid(fit1) ~ Rubber$tens,ylim=c(-80,80)) plot(resid(fit5) ~ Rubber$tens,ylim=c(-80,80)) par(mfrow=c(1,1)) |
Supponiamo di voler modellizare la dipendenza della variabile
![]() dalla variabile
dalla variabile
![]() per i dati nel dataframe
iris. I dati suggeriscono una dipendenza lineare
per i dati nel dataframe
iris. I dati suggeriscono una dipendenza lineare
plot(Petal.Length ~Petal.Width,data=iris,col=palette()[iris$Species]) lm1=lm(Petal.Length ~ Petal.Width,data=iris) cf=coef(lm1) curve(cf[1]+cf[2]*x,add=T) |
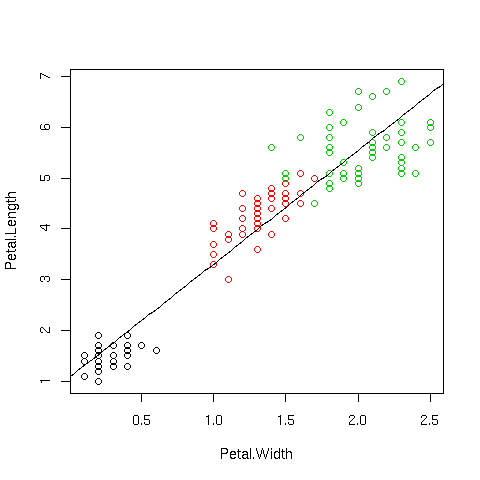
Possiamo ora cercare di migliorare il modello tenendo conto di una
eventuale dipendenza dei parametri dalla specie (si noti l'argomento
![]() della funzione
della funzione ![]() che permette di
disegnare i punti con colore diverso a seconda della specie).
che permette di
disegnare i punti con colore diverso a seconda della specie).
Le funzioni di R permettono di eseguire un fit in cui uno o più
parametri possono dipendere da una variabile categoriale (factor).
Possiamo dunque far dipendere ![]() o
o ![]() dalla specie:
dalla specie:
plot(Petal.Length ~ Petal.Width,data=iris,col=palette()[iris$Species]) # intercetta comune, pendenza dipendente dalla specie lm2=lm(Petal.Length ~ 1 + Species:Petal.Width,data=iris) cf=lm2$coefficients curve(cf[1]+cf[2]*x,add=T,col=1) curve(cf[1]+cf[3]*x,add=T,col=2) curve(cf[1]+cf[4]*x,add=T,col=3) # pendenza comune, intercetta dipendente dalla specie lm3=lm(Petal.Length ~ Species - 1 + Petal.Width,data=iris) cf=lm3$coefficients curve(cf[1]+cf[4]*x,add=T,col=1,lty=2) curve(cf[2]+cf[4]*x,add=T,col=2,lty=2) curve(cf[3]+cf[4]*x,add=T,col=3,lty=2) |
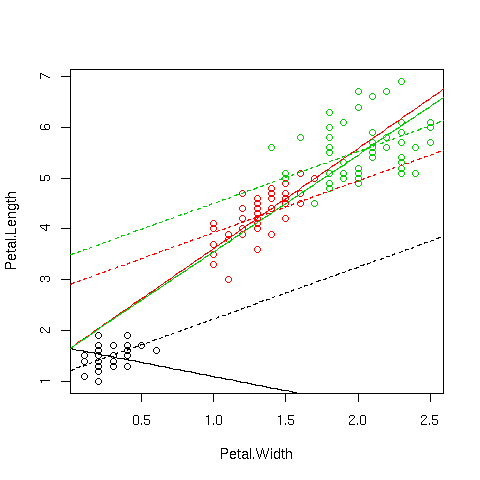
(si noti la sintassi Species - 1 per ottenere un'intercetta
![]() dipendente dalla specie. Se avessimo omesso il
termine -1, R avrebbe aggiunto un'intercetta comune e il
risultato sarebbe stato equivalente, ma i parametri del fit sarebbero
stati
dipendente dalla specie. Se avessimo omesso il
termine -1, R avrebbe aggiunto un'intercetta comune e il
risultato sarebbe stato equivalente, ma i parametri del fit sarebbero
stati
![]() .)
.)
L'analisi della varianza ci suggerisce che per entrambi i modelli
![]() e
e ![]() , l'aggiunta di 2 parametri rispetto al modello lm1 è
giustificata
, l'aggiunta di 2 parametri rispetto al modello lm1 è
giustificata
> anova(lm1,lm2) Analysis of Variance Table Model 1: Petal.Length ~ Petal.Width Model 2: Petal.Length ~ 1 + Species:Petal.Width Res.Df RSS Df Sum of Sq F Pr(>F) 1 148 33.845 2 146 25.563 2 8.282 23.65 1.267e-09 *** --- > anova(lm1,lm3) Analysis of Variance Table Model 1: Petal.Length ~ Petal.Width Model 2: Petal.Length ~ Species - 1 + Petal.Width Res.Df RSS Df Sum of Sq F Pr(>F) 1 148 33.845 2 146 20.833 2 13.011 45.591 4.137e-16 *** |
Possiamo infine far dipendere entrambi i parametri dalla specie (equivalente a fare 3 fits indipendenti per ciascuna specie)
plot(Petal.Length ~ Petal.Width,data=iris,col=palette()[iris$Species]) # pendenza e intercetta dipendente dalla specie lm4=lm(Petal.Length ~ Species - 1 + Species:Petal.Width,data=iris) cf=lm4$coefficients curve(cf[1]+cf[4]*x,add=T,col=1,lty=3) curve(cf[2]+cf[5]*x,add=T,col=2,lty=3) curve(cf[3]+cf[6]*x,add=T,col=3,lty=3) |
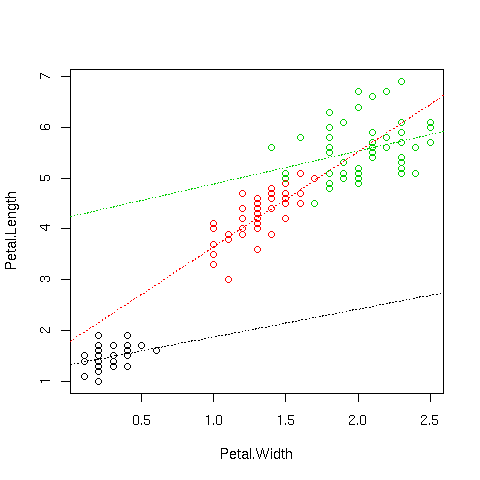
Anche in questo caso, i due parametri aggiuntivi sembrano giustificati:
> anova(lm3,lm4) Analysis of Variance Table Model 1: Petal.Length ~ Species - 1 + Petal.Width Model 2: Petal.Length ~ Species - 1 + Species:Petal.Width Res.Df RSS Df Sum of Sq F Pr(>F) 1 146 20.8334 2 144 18.8156 2 2.0178 7.7213 0.0006525 *** --- Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 |