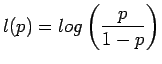
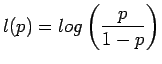
che parametrizziamo in funzione delle categorie di età, sesso, e classe di viaggio assumendo l'indipendenza dei 3 fattori:
Possiamo eliminare la ridondanza dei parametri ponendo
![]() .
.
# e' necessario modificare il dataframe per avere per ogni riga il numero di sopravvissuti
# e quello totale di ciascuna categoria
library(boot)
Tit=as.data.frame(Titanic)
c.vivi = Tit[Tit$Survived=="Yes","Freq"]
c.tutti = Tit[Tit$Survived=="No","Freq"] + c.vivi
NewTit=data.frame(Tit[Tit$Survived=="Yes",1:3],vivi=c.vivi,tutti=c.tutti)
#Eseguiamo il fit sulla frequenza vivi/tutti
logfit= glm(vivi/tutti ~ Age -1 +Sex+Class , weight=tutti,
family=binomial,data=NewTit)
summary(logfit)
|
Dal risultato notiamo come tutti i parametri siano significativamente diversi da 0.
Per ottenere la probabilità di sopravvivenza ottenuta dal fit e il suo
errore, usiamo la funzione ![]() di R. Si noti l'opzione type="response" che ci permette di ottenere direttamente i valori di
di R. Si noti l'opzione type="response" che ci permette di ottenere direttamente i valori di
![]() anzichè la quantità fittata
anzichè la quantità fittata ![]() :
:
> results=predict(logfit2,type="response",se.fit=T,newdata=NewTit) > NewTit=data.frame(NewTit, prob.fit=results$fit,dprob.fit=results$se.fit) > NewTit Class Sex Age vivi tutti prob.fit dprob.fit 17 1st Male Child 5 5 0.6649249 0.06082307 18 2nd Male Child 11 11 0.4175655 0.06533645 19 3rd Male Child 13 48 0.2511586 0.04458132 20 Crew Male Child 0 0 0.4570172 0.06370410 21 1st Female Child 1 1 0.9571141 0.01223942 22 2nd Female Child 13 13 0.8896612 0.02786822 23 3rd Female Child 14 31 0.7904463 0.04108526 24 Crew Female Child 0 0 0.9044523 0.02582406 25 1st Male Adult 57 175 0.4070382 0.03286746 26 2nd Male Adult 14 168 0.1987193 0.02474444 27 3rd Male Adult 75 462 0.1039594 0.01182252 28 Crew Male Adult 192 862 0.2254997 0.01405835 29 1st Female Adult 140 144 0.8853234 0.01704920 30 2nd Female Adult 80 93 0.7360897 0.03241455 31 3rd Female Adult 76 165 0.5661291 0.03175476 32 Crew Female Adult 20 23 0.7660538 0.02841769 |