
Il problema potrebbe essere risolto fittando l'istogramma della
distribuzione della variabile ![]() . La soluzione più generale
possibile, che sfrutta interamente l'informazione disponibile, pena un
maggiore dispendio di CPU, consiste tuttavia nello stimare i parametri
massimizzando direttamente la funzione di likelihood del nostro
campione
. La soluzione più generale
possibile, che sfrutta interamente l'informazione disponibile, pena un
maggiore dispendio di CPU, consiste tuttavia nello stimare i parametri
massimizzando direttamente la funzione di likelihood del nostro
campione
Usiamo per questo la funzione ![]() del pacchetto
del pacchetto ![]() che
permette di minimizzare
che
permette di minimizzare ![]() con vari
algoritmi di ottimizzazione. Scegliamo l'algoritmo "L-BFGS-B", che permette di definire
dei limiti per i parametri del fit. Notiamo che nel nostro caso i
parametri
con vari
algoritmi di ottimizzazione. Scegliamo l'algoritmo "L-BFGS-B", che permette di definire
dei limiti per i parametri del fit. Notiamo che nel nostro caso i
parametri
![]() e
e
![]() possono facilmente
essere ``scambiati'' (con un probabile fallimento della
minimizzazione) se non rendiamo chiara la loro interpretazione
scegliendo in modo sensato i valori di partenza e i limiti
entro i quali eseguire la minimizzazione.
possono facilmente
essere ``scambiati'' (con un probabile fallimento della
minimizzazione) se non rendiamo chiara la loro interpretazione
scegliendo in modo sensato i valori di partenza e i limiti
entro i quali eseguire la minimizzazione.
library(stats4)
geysermodel = function(x,alpha,mu1,sigma1,mu2,sigma2) {
alpha * dnorm(x,mean=mu1,sd=sigma1) +
(1-alpha) * dnorm(x,mean=mu2,sd=sigma2)
}
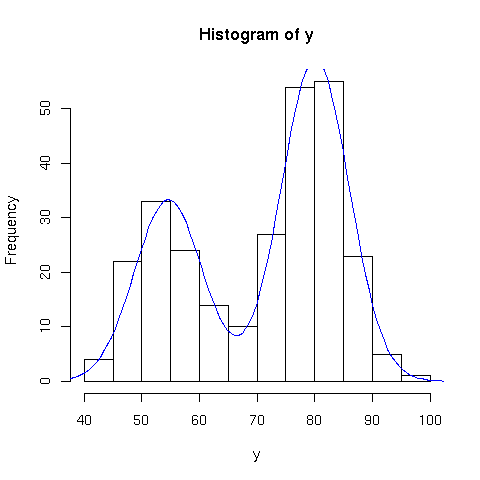
geyser.fit = function(y=faithful$waiting) {
h=hist(y)
startp=
list(alpha=0.5,mu1=55,sigma1=5 ,mu2=80,sigma2=5)
minp=c(alpha=0.1,mu1=45,sigma1=1 ,mu2=75,sigma2=1)
maxp=c(alpha=0.9,mu1=60,sigma1=10,mu2=85,sigma2=10)
llgeyser = function(alpha,mu1,sigma1,mu2,sigma2) {
-sum(log(geysermodel(y,alpha,mu1,sigma1,mu2,sigma2)) )
}
# l'argomento di mle deve essere una funzione che ritorna -logL
fit=mle(llgeyser,start=startp,lower=minp,upper=maxp,method="L-BFGS-B")
print(summary(fit))
cf=fit@coef
curve(geysermodel(x,cf[1],cf[2],cf[3],cf[4],cf[5]) * (h$breaks[2]-h$breaks[1]) * length(y),
add=T,col="blue")
list( min= fit@min , coef= cf, var=vcov(fit))
}
|
> expfit = geyser.fit()
Maximum likelihood estimation
Call:
mle(minuslogl = llgeyser, start = startp, method = "L-BFGS-B",
lower = minp, upper = maxp)
Coefficients:
Estimate Std. Error
alpha 0.3608887 0.03116472
mu1 54.6152983 0.69972712
sigma1 5.8715085 0.53739009
mu2 80.0912040 0.50459193
sigma2 5.8677196 0.40096112
-2 log L: 2068.004
Per poter eseguire un test di bontà del fit, è necessario conoscere la
distribuzione attesa di
![]() nell'ipotesi che il modello
sia corretto.
nell'ipotesi che il modello
sia corretto.
Essendo un'impresa ardua determinare analiticamente questa
distribuzione nel nostro caso (e questo vale per tutti i casi in cui
la distribuzione ![]() non sia particolarmente semplice), ricorriamo
alla simulazione di
non sia particolarmente semplice), ricorriamo
alla simulazione di ![]() esperimenti con
esperimenti con ![]() valori della nostra
variabile simulati secondo la funzione
valori della nostra
variabile simulati secondo la funzione ![]() :
:
geyser.check = function(Nsim=100) {
# inizializziamo un vettore che conterra' i valori di min(log(L)) di
# ogni esperimento simulato
outll=c()
n=length(faithful$waiting)
for (i in 1:Nsim) {
# il numero di eventi del primo tipo segue una distribuzione binomiale
# con prob. alpha
n1=rbinom(1,size=n,prob=expfit$coef["alpha"])
n2=n-n1
sample=c( rnorm(n1,mean=expfit$coef["mu1"],sd=expfit$coef["sigma1"]) ,
rnorm(n2,mean=expfit$coef["mu2"],sd=expfit$coef["sigma2"]) )
simfit=geyser.fit(sample)
outll[i]=simfit$min
}
# distribuzione ottenuta dalla simulazione:
hist(outll)
cat("min logL =",expfit$min,"\n")
# disegniamo una linea verticale in corrispondenza del valore sperimentale
lines(c(expfit$min,expfit$min),c(0,Nsim))
}
|
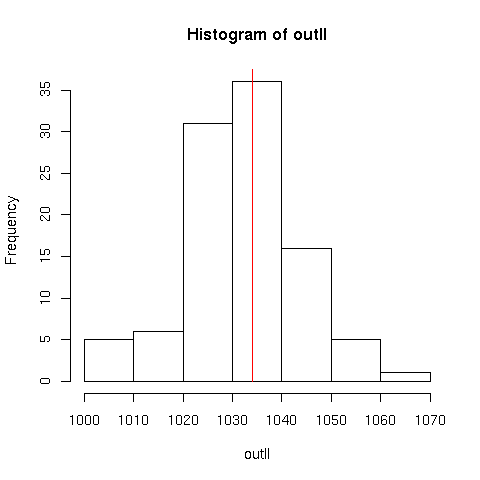
Come osserviamo dal grafico, il valore ottenuto sperimentalmente è perfettamente compatibile con il nostro modello.