
Utilizziamo la funzione ![]() di R per questa analisi di regressione
non lineare. L'argomento
di R per questa analisi di regressione
non lineare. L'argomento ![]() deve contenere un vettore con i
parametri liberi e il loro valore di partenza. Ogni parametro deve
avere il nome che viene usato nella formula del modello. Nel nostro
caso abbiamo un solo parametro che chiamiamo ``tau'':
deve contenere un vettore con i
parametri liberi e il loro valore di partenza. Ogni parametro deve
avere il nome che viene usato nella formula del modello. Nel nostro
caso abbiamo un solo parametro che chiamiamo ``tau'':
mydata=read.table('/afs/math.unifi.it/service/Rdsets/decay.rdata')
plot(signal ~ time,data=mydata)
fit=nls(signal ~ exp(-time/tau), data=mydata,
start=c(tau=2) )
lines(mydata$time,predict(fit))
|
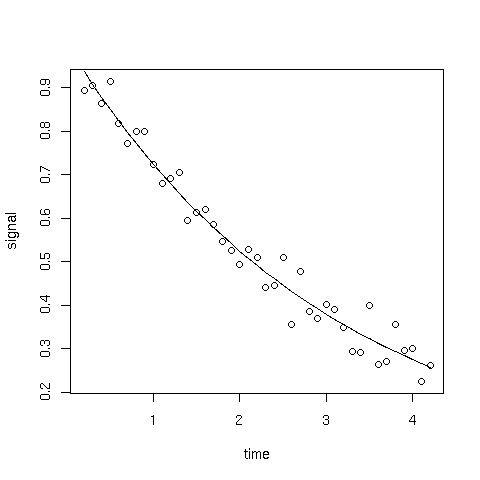
Dalla minimizzazione numerica del ![]() il fit ottiene
il fit ottiene
![]()
La variabile
N=length(mydata$signal)
tau=fit$m$getPars()[1]
alpha=0.9
dtau=sqrt(vcov(fit)[1,1])
Nsigma = qt((1+alpha)/2,df=N-1)
cat ("tau=",tau," +/- ",Nsigma*dtau,"\n")
|
L'intervallo di confidenza così ottenuto presuppone l'aver assunto una
distribuzione di probabilità gaussiana per lo stimatore
![]() ,
cosa che nel caso non lineare è rigorosamente vera solo nel limite
asintotico
,
cosa che nel caso non lineare è rigorosamente vera solo nel limite
asintotico
![]() .
.
Per verificare questa approssimazione, confrontiamo il profilo del
![]() in funzione di
in funzione di ![]() , con la predizione gaussiana
, con la predizione gaussiana
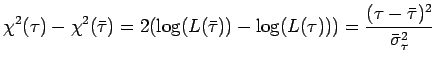
sigmay2=deviance(fit)/(N-1)
chi2.taufit = function(tau) {
out=c()
for (i in 1:length(tau) ) {
out[i]=sum( ( mydata$signal - exp(-mydata$time/tau[i]) )^2 )/sigmay2
}
out
}
# profilo effettivo in nero
curve(chi2.taufit(x),tau-3*dtau,tau+3*dtau,
xlab=expression(tau),ylab=expression(chi^2))
# predizione approssimato in rosso
curve((N-1)+(x-tau)^2/dtau^2,add=T,col="red")
|
Il plot dimostra la validità dell'approssimazione. Se infatti
volessimo migliorare la stima dell'intervallo di confidenza,
calcolando i valori di ![]() per cui
per cui
![]() , otterremmo
, otterremmo
![]() al 90 % C.L.
al 90 % C.L.
con una variazione della stima degli errori di circa il 2 %.

Ripetiamo il procedimento con due parametri liberi
mydata=read.table('/afs/math.unifi.it/service/Rdsets/decay.rdata')
plot(signal ~ time,data=mydata)
fit=nls(signal ~ I0*exp(-time/tau), data=mydata,
start=c(I0=1,tau=2) )
print(summary(fit))
lines(mydata$time,predict(fit))
|
da cui si ottiene
Parameters:
Estimate Std. Error t value Pr(>|t|)
I0 1.00321 0.01757 57.09 <2e-16 ***
tau 3.08820 0.09752 31.67 <2e-16 ***
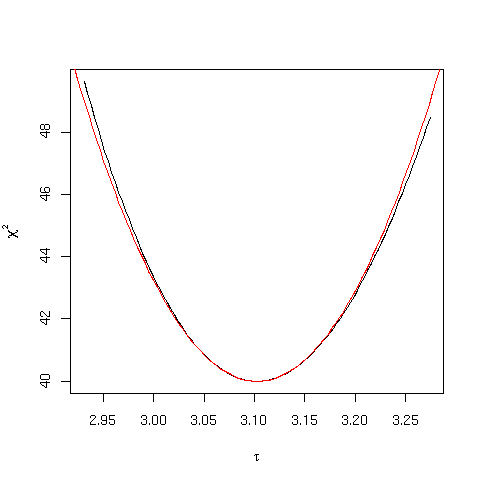
In questo caso, l'approssimazione gaussiana prevede che il profilo del
![]() sia dato dal logaritmo di una gaussiana bivariata
sia dato dal logaritmo di una gaussiana bivariata
![$\displaystyle \chi^2(\tau,I_0) - \chi^2(\bar{\tau},\overline{I_0}) = (\theta -
...
...rho (\tau-\bar{\tau})(I_0-\overline{I_0})}{\sigma_{\tau}\sigma_{I_0}}
\right]
$](img242.gif)
N=length(mydata$signal)
theta=fit$m$getPars()
V.theta=vcov(fit)
dtau=sqrt(diag(vcov(fit)))
sigmay2=deviance(fit)/(N-2)
chi2.taufit2 = function(I0,tau) {
out=matrix(nrow=length(I0),ncol=length(tau))
for (i in 1:length(I0) ) {
for (j in 1:length(tau) ) {
out[i,j]=sum( ( mydata$signal - I0[i]*exp(-mydata$time/tau[j]) )^2 )/sigmay2
}
}
out
}
pred.chi2.taufit2 = function(I0,tau) {
out=matrix(nrow=length(I0),ncol=length(tau))
for (i in 1:length(I0) ) {
for (j in 1:length(tau) ) {
xy=c(I0[i],tau[j])
out[i,j]= (N-2) + t(xy-theta) %*% solve(V.theta) %*% (xy-theta)
}
}
out
}
nsigma=c(1,2,3)
x=seq(0.95,1.05,0.001)
y=seq(2.5,3.5,0.005)
# profilo effettivo in nero
contour(x,y, chi2.taufit2(x,y) , levels = (N-2) + nsigma^2,
xlab=expression(I0),ylab=expression(tau))
# predizione approssimata in rosso
contour(x,y, pred.chi2.taufit2(x,y) , levels =(N-2) + nsigma^2,
col="red",add=T )
|
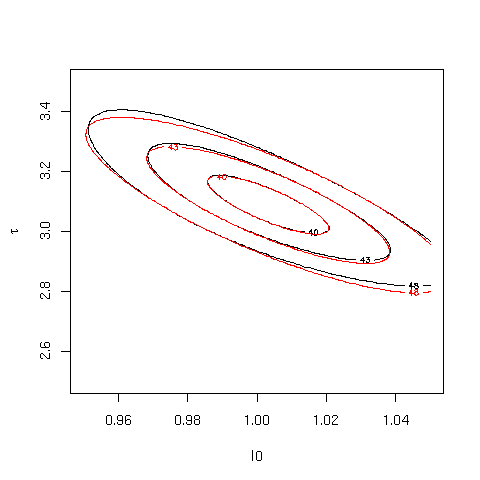

Essendo il modello non lineare, utilizziamo la funzione nls() con i tre parametri liberi
library(MASS)
plot(Weight ~ Days,data=wtloss)
theta.start=c(W.0=100, W.e=100,D.half=200)
fit=nls(Weight ~ W.0 + W.e * 2^(-Days/D.half) , data=wtloss, start=theta.start)
print(summary(fit))
lines(wtloss$Days,predict(fit),col="blue")
# matrice di correlazione fra i parametri
Vcor = cov2cor(vcov(fit))
rho = Vcor["W.0","W.e"]
ngl=length(wtloss$Weight)-3
drho = (1-rho^2)/sqrt(ngl)
cat("rho(W.0,W.e)=",round(rho,3)," +/- ",round(drho,3),"\n")
|
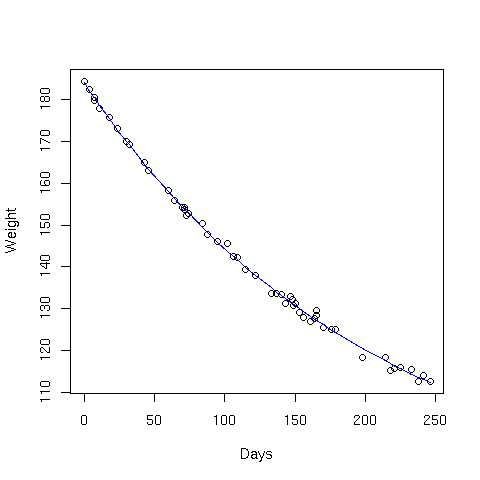
Da cui otteniamo
Parameters:
Estimate Std. Error t value Pr(>|t|)
W.0 81.374 2.269 35.86 <2e-16 ***
W.e 102.684 2.083 49.30 <2e-16 ***
D.half 141.910 5.295 26.80 <2e-16 ***
rho(W.0,W.e)= -0.989 +/- 0.003

Supponendo che la deviazione standard di ogni misura sia determinata
dalla precisione della bilancia
![]() Kg, possiamo calcolare il
Kg, possiamo calcolare il
![]() del fit ed eseguire un test dell'ipotesi che segua una
distribuzione di Pearson con
del fit ed eseguire un test dell'ipotesi che segua una
distribuzione di Pearson con ![]() gradi di libertà:
gradi di libertà:
> chi2=deviance(fit)/0.5^2
> cat("chi2=",chi2," con ",ngl," g.d.l. : p-value=",
1-pchisq(chi2,df=ngl),"\n")
chi2= 156.9788 con 49 g.d.l. : p-value= 3.100853e-13
|
Evidentemente l'errore nella misura non basta a spiegare la varianza dei residui del fit, che probabilmente è dominata da fluttuazioni fisiologiche del peso, e magari da qualche strappo alla dieta!