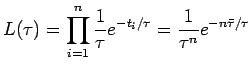

La funzione di likelihood è
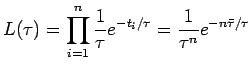
Lo stimatore di massima verosimiglianza
![]() si ottiene da
si ottiene da
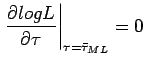
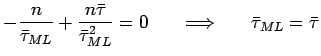
Lo stimatore ML coincide dunque con la media aritmetica che è uno
stimatore corretto del valore atteso ![]() ed ha varianza
ed ha varianza
L'informazione di Fisher è
![$\displaystyle I(\tau) = E\left( \left( \frac{\partial \log{L}}{\partial \tau}
\...
...tau^2}\left(
\frac{\tau^2}{n}+\tau^2 \right) -2 +1 \right] = \frac{n}{\tau^2}
$](img61.gif)
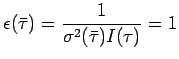
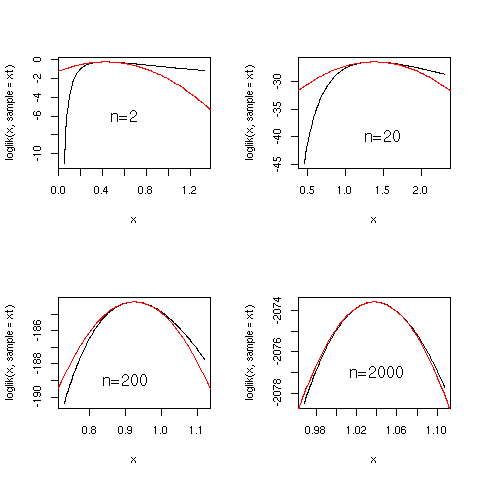
Con il seguente codice verifichiamo che il limite asintotico (
![]() )
della funzione di lihelihood è
)
della funzione di lihelihood è
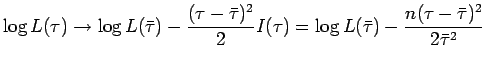
loglik = function (tau,sample) {
n=length(sample)
etau=mean(sample)
-n*(log(tau)+etau/tau)
}
seelik = function(n=1000) {
xt=rexp(n)
etau=mean(xt)
detau=etau/sqrt(n)
cat("tau estimate is ",etau," +/- ",detau,"\n")
curve(loglik(x,sample=xt),max(etau-3*detau,0.05),etau+3*detau)
curve(loglik(etau,sample=xt)-(x-etau)^2/(2*detau^2),add=T,col="red")
}
par(mfrow=c(2,2))
seelik(2)
seelik(20)
seelik(200)
seelik(2000)
|

Parametrizziamo la distribuzione di probabilità della variabile misurata, l'energia x:
Una prima stima dei parametri ![]() e
e ![]() può essere ottenuta
stimando il valore atteso e la varianza della distribuzione:
può essere ottenuta
stimando il valore atteso e la varianza della distribuzione:
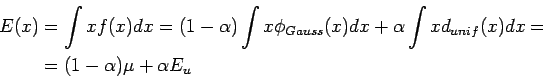 |
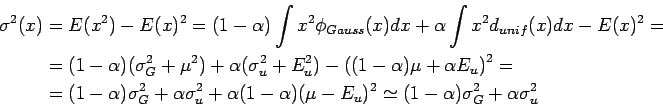 |
Ricaviamo dunque gli stimatori
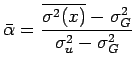
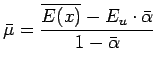
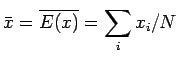
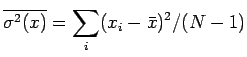
Gli errori standard su queste stime saranno:
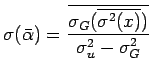
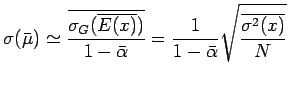
dove abbiamo trascurato l'incertezza su
![]() nell'espressione di
nell'espressione di
![]() e
e
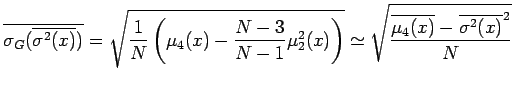
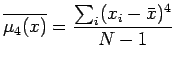
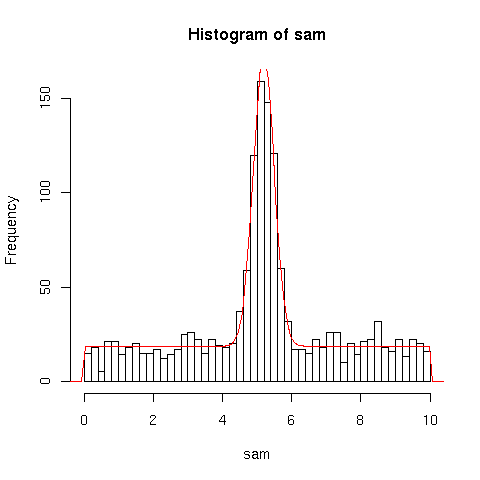
Stimiamo i valori numerici e verifichiamo graficamente il risultato:
gaussconfondo= function(x,alpha,mu,s1=0.3) {
(1-alpha)*dnorm(x,mean=mu,sd=s1)+alpha*dunif(x,min=0,max=10)
}
> sam=scan("gaussconfondo.rdata")
> n=length(sam)
> s1=0.3
> myalpha=(var(sam)-s1^2)/(25/3-s1^2)
> mu4=mean((sam-mean(sam))^4)
> dvar=sqrt((mu4-var(sam)^2)/n)
> mydalpha=dvar/(25/3-s1^2)
> mymu=(mean(sam)-5*myalpha)/(1-myalpha)
> mydmu = sqrt(var(sam)/(n*(1-myalpha)^2))
> cat ("stima di alpha: ",myalpha," +/- ",mydalpha,"\n")
stima di alpha: 0.5873126 +/- 0.02139240
> cat ("stima di mu: ",mymu," +/- ",mydmu,"\n")
stima di mu: 5.433121 +/- 0.1383849
hist(sam,breaks=seq(0.,10.,0.2))
curve(gaussconfondo(x,alpha=myalpha,mu=mymu)*n*0.2,add=T,col="red")
|
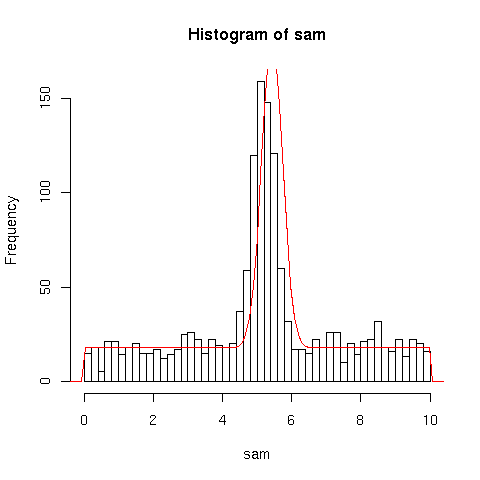
E' evidente che la stima di ![]() , seppur corretta entro l'errore
stimato, può essere migliorata. Questo non è
sorprendente dal momento che, usando la media aritmetica, abbiamo
utilizzato con lo stesso peso tutti gli eventi, anche quelli del fondo
che non portano alcuna informazione su
, seppur corretta entro l'errore
stimato, può essere migliorata. Questo non è
sorprendente dal momento che, usando la media aritmetica, abbiamo
utilizzato con lo stesso peso tutti gli eventi, anche quelli del fondo
che non portano alcuna informazione su ![]() ma aggiungono solo
``rumore'' alla nostra stima. Il metodo di ML ci
fornisce invece uno stimatore che estrae dai dati il massimo
dell'informazione. Ci aspettiamo inoltre che, vista la statistica
relativamente elevata del campione, lo stimatore abbia alta efficienza
e bias trascurabile.
ma aggiungono solo
``rumore'' alla nostra stima. Il metodo di ML ci
fornisce invece uno stimatore che estrae dai dati il massimo
dell'informazione. Ci aspettiamo inoltre che, vista la statistica
relativamente elevata del campione, lo stimatore abbia alta efficienza
e bias trascurabile.
Calcoliamo il massimo
della funzione di likelihood
numericamente minimizzando la quantità ![]() . In generale,
dovremmo trovare il minimo variando simultaneamente i due parametri
. In generale,
dovremmo trovare il minimo variando simultaneamente i due parametri
![]() e
e ![]() . Tuttavia ci aspettiamo che la correlazione fra i due
parametri sia piccola, poichè i parametri descrivono caratteristiche
diverse del campione. E' dunque ragionevole stimare un parametro alla
volta, ed eventualmente raffinare la stima procedendo in modo
iterativo:
. Tuttavia ci aspettiamo che la correlazione fra i due
parametri sia piccola, poichè i parametri descrivono caratteristiche
diverse del campione. E' dunque ragionevole stimare un parametro alla
volta, ed eventualmente raffinare la stima procedendo in modo
iterativo:
loglikgfa = function(a,sample,m) {
out=c()
for (i in 1:length(a)) {
out[i] = -sum(log(gaussconfondo(sample,alpha=a[i],mu=m)))
}
out
}
loglikgfmu = function(m,sample,a) {
out=c()
for (i in 1:length(m)) {
out[i] = -sum(log(gaussconfondo(sample,alpha=a,mu=m[i])))
}
out
}
# prima stima di mu e alpha
curve(loglikgfmu(x,sam,myalpha),5.15,5.25)
# possiamo stimare visivamente dal grafico
# mu_ML = 5.192 +/- 0.016
curve(loglikgfa(x,sam,mymu),0.5,0.7)
# possiamo stimare visivamente dal grafico
# alpha_ML = 0.650 +/- 0.015
# seconda iterazione
curve(loglikgfmu(x,sam,0.650),5.17,5.21,xlab=expression(mu))
curve(loglikgfa(x,sam,5.192),0.55,0.65,xlab=expression(alpha))
|
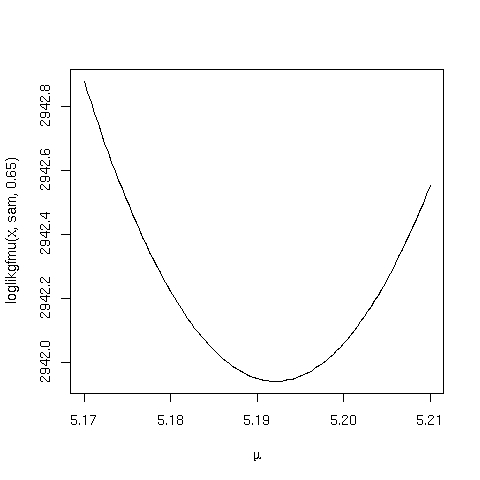
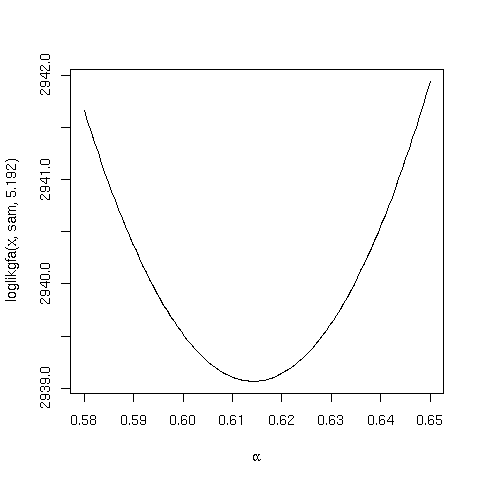
Dai grafici possiamo stimare, valutando i valori per cui ![]() differisce di 1/2 dal minimo:
differisce di 1/2 dal minimo:
Una ulteriore iterazione non produce variazioni rivelanti. Come ci si
poteva aspettare, la precisione della nostra stima è decisamente
migliorata, in particolare per ![]() . Notiamo anche come il profilo di
. Notiamo anche come il profilo di
![]() sia parabolico, confermandoci che siamo prossimi al
limite asintotico in cui gli stimatori di ML sono corretti ed
efficienti.
sia parabolico, confermandoci che siamo prossimi al
limite asintotico in cui gli stimatori di ML sono corretti ed
efficienti.
In effetti, il campione considerato è stato simulato con i valori
![]() ,
,
![]() , in perfetto accordo con le nostre stime.
, in perfetto accordo con le nostre stime.
Con un maggior dispendio di CPU avremmo potuto minimizzare la
funzione in due dimensioni utilizzando l'apposita funzione di R
![]() , che permette di scegliere fra vari algoritmi di
minimizzazione. La procedura è la seguente:
, che permette di scegliere fra vari algoritmi di
minimizzazione. La procedura è la seguente:
# e' necessario specificare i valori di partenza dei parametri e
# gli intervalli entro cui cercare il minimo
minusll = function(mu=5.4,alpha=0.65) {
-sum(log(gaussconfondo(sam,alpha=alpha,mu=mu)))
}
> library(stats4)
> mle(minusll,method="L-BFGS-B",lower=c(5.0,0.5),upper=c(5.4,0.7)) -> fit
> summary(fit)
Maximum likelihood estimation
Call:
mle(minuslogl = minusll, method = "L-BFGS-B", lower = c(5, 0.5),
upper = c(5.4, 0.7))
Coefficients:
Estimate Std. Error
mu 5.1918609 0.01579727
alpha 0.6142559 0.01498571
-2 log L: 5878.132
|
Come previsto, abbiamo ritrovato i valori ottenuti ``manualmente''.
La funzione ![]() è in grado di stimare la matrice
varianza/covarianza dei due stimatori valutando numericamente
è in grado di stimare la matrice
varianza/covarianza dei due stimatori valutando numericamente
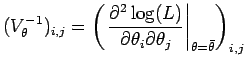
> vcov(fit)
mu alpha
mu 2.495537e-04 1.617870e-06
alpha 1.617870e-06 2.245715e-04
# in termini di coefficienti di correlazione:
> cov2cor(vcov(fit))
mu alpha
mu 1.000000000 0.006834148
alpha 0.006834148 1.000000000
|
Otteniamo dunque un coefficiente di correlazione inferiore all'1 %.
Verifichiamo infine graficamente l'accordo fra i dati e la miglior stima ottenuta:
hist(sam,breaks=seq(0.,10.,0.2)) curve(gaussconfondo(x,alpha=0.614,mu=5.192)*n*0.2,add=T,col="red") |