

Come abbiamo visto, la miglior stima di ![]() per la distribuzione
esponenziale è data dalla media aritmetica, in questo caso l'unico
valore osservato
per la distribuzione
esponenziale è data dalla media aritmetica, in questo caso l'unico
valore osservato ![]() .
Per ottenere la soluzione esatta dell'intervallo di confidenza col
metodo di Neyman, dobbiamo
calcolare i quantili
.
Per ottenere la soluzione esatta dell'intervallo di confidenza col
metodo di Neyman, dobbiamo
calcolare i quantili
![]() e
e
![]() come
funzione di
come
funzione di ![]() risolvendo l'equazione
risolvendo l'equazione
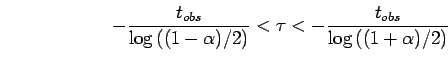
Si noti che l'intervallo è asimmetrico, poichè la miglior stima resta
![]() .
.
La stima ``standard'', che coincide con la soluzione esatta solo se la distribuzione dello stimatore è gaussiana (cosa evidentemente falsa in questo caso), è
> alpha=0.9 > qnorm((1+alpha)/2) [1] 1.644854
Con il metodo di massima verosimiglianza l'intervallo è stimato risolvendo l'equazione
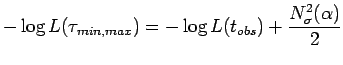
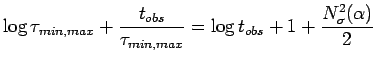
mloglike =function(tau=1,tobs) {
log(tau)+tobs/tau
}
nsig = function(alpha) {
qnorm((alpha+1)/2)
}
confneyman = function(tobs,alpha) {
c( -tobs/log((1-alpha)/2) ,
tobs,
-tobs/log((1+alpha)/2))
}
confstd = function(tobs,alpha) {
terr = nsig(alpha) * tobs
c( tobs - terr,
tobs,
tobs + terr)
}
confll = function(tobs,alpha) {
# soluzione ML: partendo dal massimo, ci spostiamo di una quantita'
# piccola (rispetto all'errore) step, fino a trovare il valore per cui
# loglik varia di nsig^2/2
deltal=(nsig(alpha))^2/2
llmin=mloglike(tobs,tobs)
step = (nsig(alpha)*tobs)/20
tminll=tobs
while (mloglike(tminll,tobs) < llmin+deltal) {
tminll = tminll - step
# protezione contro log(numero negativo)
if(tminll<0) break
}
tmaxll=tobs
while(mloglike(tmaxll,tobs) < llmin+deltal) {
tmaxll = tmaxll + step
}
c( tminll,
tobs,
tmaxll)
}
seeconf = function(alpha=0.9,tobs=0.6) {
# disegna il diagramma di Neyman
curve(x*1,0,10*tobs,xlab=expression(tau),ylab=expression(t_obs),xlim=c(0,20*tobs))
curve(-x*log((1-alpha)/2),add=T,col="blue",0,10*tobs)
curve(-x*log((1+alpha)/2),add=T,col="blue",0,20*tobs)
# soluzione esatta
ney = confneyman(tobs,alpha)
cat("tau is ",ney[2]," - ",(ney[2]-ney[1])," + ",(ney[3]-ney[2]),"\n")
lines(c(ney[1],ney[3]),c(ney[2],ney[2]),col="red",type="b")
# soluzione standard
std = confstd(tobs,alpha)
cat("tau standard is ",std[2]," +/- ",std[2]-std[1],"\n")
lines(c(std[1],std[3]),c(std[2]+0.1,std[2]+0.1),col="magenta",type="b")
# soluzione ML
ll = confll(tobs,alpha)
cat("taull is ",ll[2]," - ",ll[2]-ll[1]," + ",ll[3]-ll[2],"\n")
lines(c(ll[1],ll[3]),c(ll[2]-0.1,ll[2]-0.1),col="green",type="b")
}
> seeconf()
tau is 0.6 - 0.3997151 + 11.09744
tau standard is 0.6 +/- 0.9869122
taull is 0.6 - 0.464758 + 5.112338
|
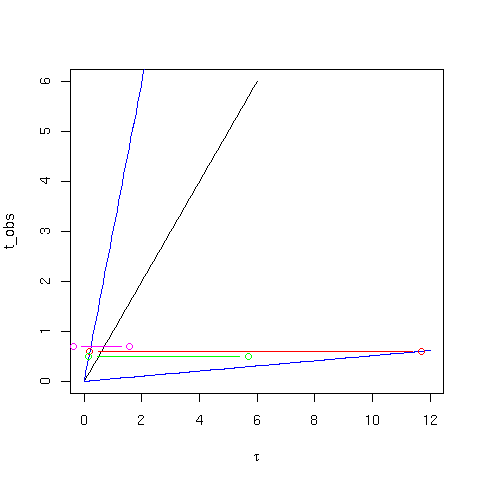
Per quantificare la differenza fra il significato dei tre intervalli,
possiamo calcolarne il ``ricoprimento'', ovvero l'effettivo livello di
confidenza. E' necessario per questo simulare un gran numero di
esperimenti e contare quante volte l'intervallo ottenuto contiene il
valore vero di ![]() usato nella simulazione. Per l'intervallo
ottenuto col metodo di Neyman ci aspettiamo naturalmente di trovare un
ricoprimento pari ad
usato nella simulazione. Per l'intervallo
ottenuto col metodo di Neyman ci aspettiamo naturalmente di trovare un
ricoprimento pari ad ![]() .
.
coverage = function(n=1000,tau=1,alpha=0.9) {
# simuliamo gli n esperimenti, si noti che il valore di tau
# e' ininfluente sul risultato
xx=rexp(n,rate=1/tau)
nokney=0
nokst=0
nokll=0
for (i in 1:n) {
tobs=xx[i]
# intervallo di neyman
ney = confneyman(tobs,alpha)
if(tau >= ney[1] & tau <= ney[3]) {
nokney=nokney+1
}
# intervallo standard
std = confstd(tobs,alpha)
if(tau >= std[1] & tau <= std[3]) {
nokst=nokst+1
}
# intervallo ML
ll = confll(tobs,alpha)
if(tau >= ll[1] & tau <= ll[3]) {
nokll=nokll+1
}
}
# calcoliamo il ricoprimento e il suo errore (binomiale)
covney=nokney/n
dcovney=sqrt(covney*(1-covney)/n)
cat("coverage Neyman =",covney," +/- ",dcovney,"\n")
covst=nokst/n
dcovst=sqrt(covst*(1-covst)/n)
cat("coverage standard=",covst," +/- ",dcovst,"\n")
covll=nokll/n
dcovll=sqrt(covll*(1-covll)/n)
cat("coverage ML=",covll," +/- ",dcovll,"\n")
}
> coverage(n=20000,alpha=0.9)
coverage Neyman = 0.9004 +/- 0.002117544
coverage standard = 0.6813 +/- 0.003294923
coverage ML = 0.87505 +/- 0.002338135
|